基因组变异库GVM(Genome Variation Map)全新升级
近日,国家生物信息中心全新升级的多物种基因组变异库GVM(Genome Variation Map)正式上线。该研究内容以“Genome Variation Map: a platform for the analysis and integration of genomic variation”为题在学术期刊Nucleic Acids Research在线发表。
随着测序技术的快速发展,越来越多物种的基因组被高质量解析,不同物种及其群体的全基因组变异数据呈现指数式增长,为遗传多样性评估、群体进化分析、功能基因挖掘、分子育种和疾病相关变异研究等提供了坚实的数据支撑。自2017年上线以来,GVM持续推进多物种变异组学数据的标准化审编分析、规范汇交、开放共享与安全管理,已成为我国遗传变异研究的重要基础设施。
数据整合方面,GVM新版本收录了约21.4亿个经标准化分析流程鉴定的变异位点,覆盖包括人类、动物、植物及病原微生物等73个物种;归档了来自全球180家科研机构汇交的898个项目遗传变异数据集,覆盖了79个物种的764,837个生物学样本。此外,GVM构建了一套规范化的变异数据整合流程,通过系统整合来自包括“自主审编分析”、“用户提交”和“欧洲变异档案库(European Variation Archive,EVA)”三种来源的变异数据,提供全面的参考变异数据集。这些数据将成为物种遗传变异特征识别、功能基因鉴定以及群体遗传结构研究的高质量的数据资源。
功能注释方面,GVM新增了有害变异注释与群体遗传学选择信号分析模块。模块为27种动植物提供约1,166万条有害突变注释信息,并为8个主要农作物提供选择信号分析结果查询,为研究变异对生物功能的影响、群体适应性和进化压力提供了数据支撑,推动分子育种与进化机制等研究的深入开展。
分析工具方面,GVM上线了单倍型分析HapView和基因组版本转换VersionMap在线工具。其中,HapView提供了多种动植物群体的单倍型统计、展示与下载功能,方便使用者获取单体型分布特征;VersionMap可实现不同基因组组装版本间变异位点的精准对应,大幅提升跨版本数据使用效率。
国家生物信息中心助理研究员白雪、硕士研究生谢海霞、工程师罗洪和硕士研究生任思伟为本文共同第一作者,宋述慧研究员和田东梅高级工程师为通讯作者。该研究得到了农业农村部国家科技重大专项、中国科学院网络安全和信息化专项、中国科学院战略性先导科技专项、国家重点研发计划以及国家自然科学基金的资助。
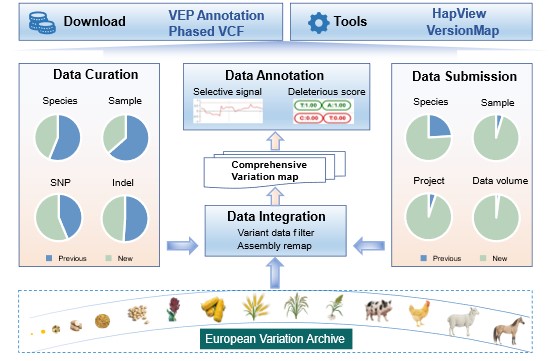
GVM 更新概览






