国家生物信息中心合作开发细胞身份鉴定新型AI引擎
随着单细胞和空间组学技术的快速发展,公开可共享数据量已突破亿级大关。然而,技术平台产生的差异、复杂疾病状态、跨物种研究带来的批次效应和离群细胞(Out-of-Distribution Cell,OOD细胞)等,对数据解读构成巨大挑战。面对动辄百万规模的OOD细胞,依赖“先聚类、后注释”的传统分析方法已显现出明显的局限性,难以快速、精准且可解释地将这些“身份不明”的细胞映射到日益完善的参考细胞图谱上。如何高效实现细胞的数字化表征、整合与解析,已成为一个关键的瓶颈问题,严重制约着单细胞数据在跨大规模人群队列研究、多模态信息整合以及物种间保守性探索等核心方向上的潜力。
近日,国家生物信息中心计算生物学部蒋岚团队在Genome Biology 期刊发表了题为CellMemory: hierarchical interpretation of out-of-distribution cells using bottlenecked transformer的研究论文,研发了一款高效、泛化且可解释的有监督细胞表征和解析模型CellMemory。该模型受全局工作空间理论(Global Workspace Theory, GWT)启发,对传统Transformer架构进行改造,植入低维记忆空间“Memory Space”,通过Cross-Attention机制将高维基因特征压缩、竞争、广播,提高计算效率3-5倍,显著增强模型泛化能力,无需预训练即可实现单细胞数据跨平台、物种整合。同时,记忆空间为CellMemory带来分层式“可读窗口”。L1 (Gene Level)为面对特定细胞,研究者可知单个基因对目标细胞表征的贡献分数;L2 (Gene Program Level)为模型在记忆空间中,自动归纳协调的共表达/共调控模式。多层可解释性为理解模型决策逻辑,探索表型关联细胞状态提供了可靠解决方案,即“高准确性 + 强可解释性”。
研究团队将CellMemory与3个单细胞基础大模型、16个任务专用模型在1500万细胞上进行比较。基准评测结果显示,CellMemory在人群尺度的单细胞数据整合、超高分辨率细胞状态注释等任务中均取得了State-of-the-Art级别的表现。面对59张MERFISH小鼠脑空间组学切片(4百万细胞、338个细胞亚群),相较基于传统transformer架构预训练的单细胞基础大模型, CellMemory在95%的空间切片上取得领先的注释表现,准确率较传统机器学习方法提升30%,证明CellMemory出色的泛化能力。
当前,将疾病细胞与健康细胞比对仍然是巨大挑战。得益于准确与可解释的细胞表征,研究团队进一步利用CellMemory在多个癌症队列单细胞图谱中解析疾病复杂性。例如在肺腺癌队列中,模型基于参考图谱定位到MSLN+ CAPN8+ 的肺泡2型过渡态细胞,并观测到其显著的拷贝数变异,提示肺腺癌可能利用肺泡2型细胞可塑性获得侵袭能力。在混合表型急性白血病、髓母细胞瘤等数据中,模型基于健康参考图谱揭示了不同患者潜在的异质性起源,为耐药和预后研究提供了高分辨率数据解析基础,展示出CellMemory在离群细胞推断场景中的强大表征能力。
综上,从“序列搜索”到“亚群搜索”,参考映射正在重塑单细胞数据分析的技术范式。凭借强大的泛化能力与高效的计算效率,CellMemory有望成为覆盖病理、时空和物种等多维度细胞参考图谱建设与临床精准诊疗的关键引擎。
上述工作由国家生物信息中心蒋岚团队和多家单位合作完成。蒋岚研究员、新加坡国立大学刘钿渤教授、加拿大麦吉尔大学李岳教授为本文的共同通讯作者。蒋岚团队博士研究生王弃非,加拿大麦吉尔大学博士生朱赫为文章的并列第一作者。清华大学张学工、斯坦福大学James Zou,博德研究所Manolis Kellis教授对本项目亦有贡献。该研究得到国家重点研发计划、中国科学院先导专项、中国科学院全球共性挑战专项等项目的资助。
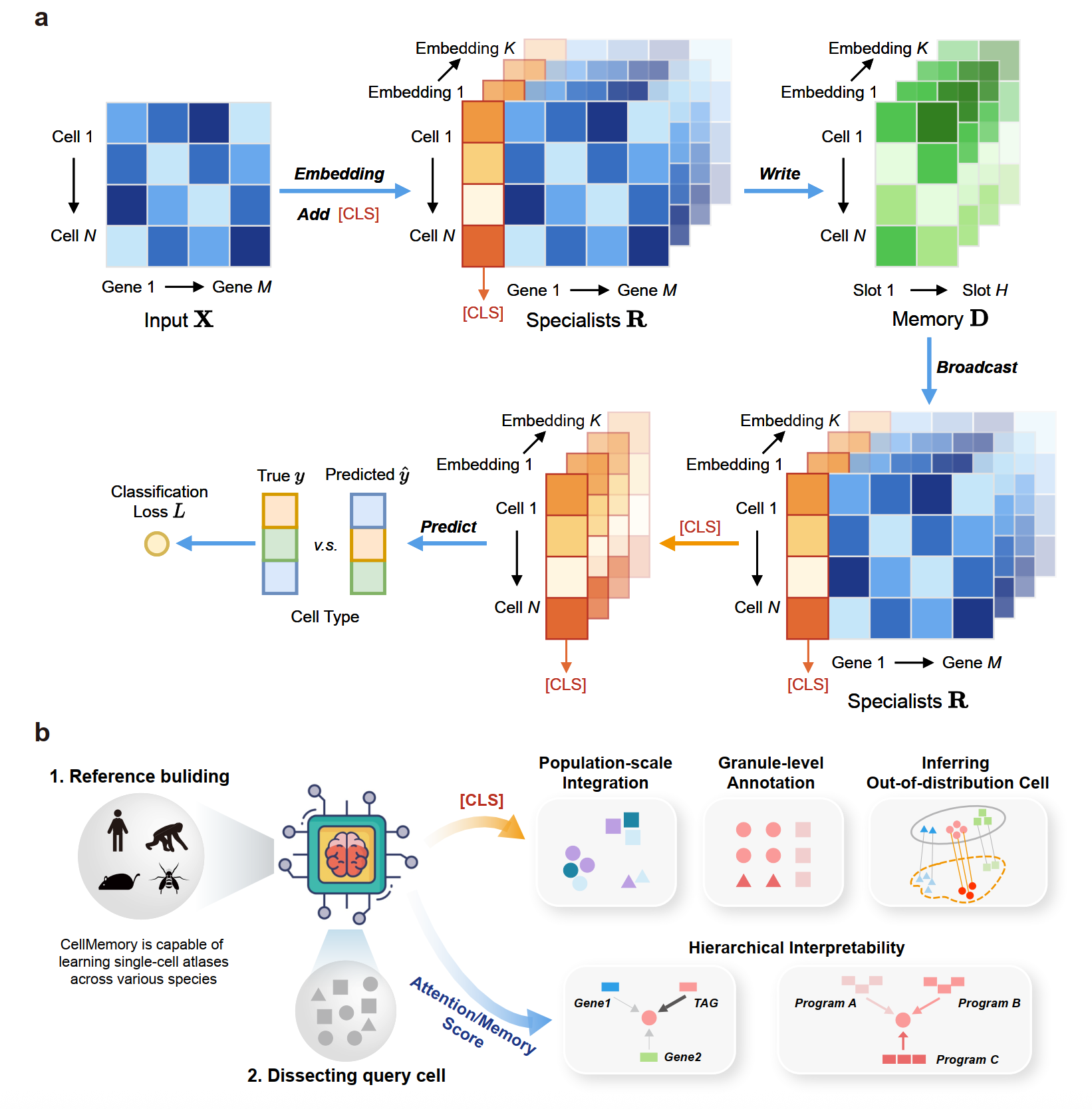
CellMemory模型架构与应用场景






